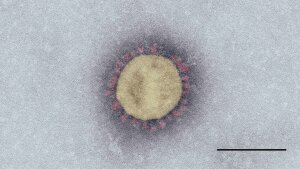
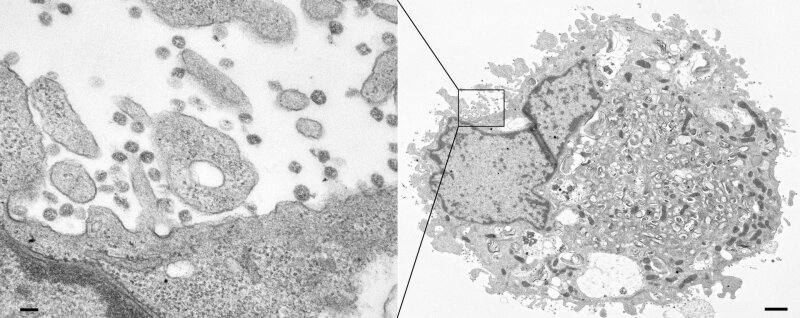
Elektronenmikroskopische Aufnahme eines SARS-Coronavirus-2. Maßstab: 100 nm.
Abbildung: Tobias Hoffmann, Michael Laue/RKISARS-CoV-2 (engl: severe acute respiratory syndrome coronavirus 2) ist Ende 2019 erstmals in China aufgetreten. Der oft auch einfach als »neuartiges Coronavirus« bezeichnete Erreger ist mit dem SARS-Virus eng verwandt (SARS-CoV-1), das 2002/2003 eine Pandemie auslöste. Auch mit dem Virus MERS-CoV besteht eine nahe Verwandschaft, das sich seit 2012 vor allem auf der Arabischen Halbinsel verbreitet hat und ebenso schwere Atemwegserkrankungen hervorruft.
Die Viruspartikel von SARS-CoV-2 haben eine kugelige Gestalt und sind im Schnitt knapp 100 Nanometer groß. Ihre Oberfläche ist mit etwa 10 Nanometer großen Spikes übersät, denen sie ihren Namen verdanken (»Corona« bedeutet Krone). Das Genom von SARS-CoV-2 und anderen Coronaviren besteht aus einzelsträngiger RNA. Es enthält knapp 30.000 Nukleotide (Kettenbausteine), die Informationen zum Bau von zehn Proteinen kodieren.
Wie Forschungsteams weltweit arbeiten seit Anfang des Jahres auch Wissenschaftlerinnen und Wissenschaftler der Jenaer Universitätsmedizin intensiv und eingebunden in interdisziplinäre Netzwerke an der Erforschung von SARS-CoV-2 und der von diesen Viren verursachten Erkankung COVID-19. Hier stellen wir exemplarisch einige Jenaer Projekte vor:
Sequenzierung von SARS-CoV-2-Patientenproben
Blick ins Genom von Coronaviren
Um festzustellen, ob sich jemand mit SARS-CoV-2 infiziert hat, wird ein Rachen- oder Nasenabstrich gemacht und das darin befindliche Virenerbgut vervielfältigt. Anschließend lassen sich definierte Genabschnitte, die für das Virus spezifisch sind, identifizieren und es kann zweifelsfrei festgestellt werden, ob in der Patientenprobe Viruspartikel vorhanden sind oder nicht.
Doch Coronavirus ist nicht gleich Coronavirus. Wie bei höheren Organismen gibt es auch im Genom von Viren Variationen. Außerdem verändert sich die Erbinformation im Laufe der Zeit durch Mutationen. Um solche genetischen Veränderungen bei SARS-CoV-2 aufzuspüren, werden einzelne Virusgenome, etwa aus Patientenproben, komplett sequenziert — also nicht nur die zur Identifizierung notwendigen Abschnitte, sondern die Abfolge der gesamten genetischen Informationen entschlüsselt. »Ziel solcher Sequenzanalysen ist es, die Verwandtschaftsstruktur von Coronaviren analysieren und so Rückschlüsse auf deren Herkunft ziehen zu können«, sagt Dr. Oliwia Makarewicz. Die Leiterin des Forschungslabors des Instituts für Infektionsmedizin und Krankenhaushygiene und ihr Team sind Teil des nationalen Konsortiums NFDI4MicrobiotaExterner Link, das in Deutschland isolierte SARS-CoV-2-Genome sequenziert. Gemeinsam mit dem Team um Bioinformatikerin Prof. Dr. Manja Marz vom Europäischen Virus-Bioinformatik-ZentrumExterner Link der Universität Jena analysieren die Forscherinnen und Forscher seit März regelmäßig die im Uniklinikum anfallenden Proben von COVID-19-Patienten. Dafür nutzen sie das MinION System – ein kleines mobiles Sequenziergerät, das, kaum größer als ein USB-Stick, direkt an einen Laptop oder PC angeschlossen werden kann.
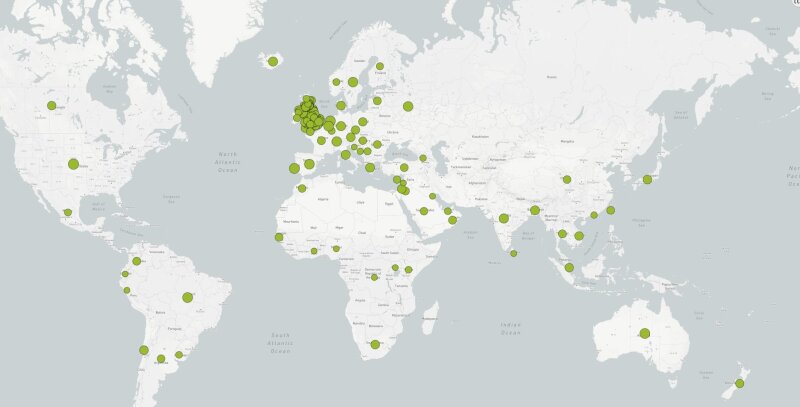
Weltweites Vorkommen der SARS-CoV-2 Lineage B.1.1. Diese Abstammungslinie ist vor allem in Europa anzutreffen.
Illustration: Erstellt mit https://pangolin.cog-uk.io/ und https://microreact.org/ am 14.07.2020»Insgesamt haben wir damit seit März etwas über 50 SARS-CoV-2-Proben sequenziert«, berichtet Dr. Christian Brandt aus dem Forschungsteam. Dabei handelt es sich vor allem um Proben, die im Jenaer Klinikum selbst entnommen worden sind. Aber auch Proben aus anderen Thüringer Kliniken und ambulanten Labors aus dem gesamten Bundesgebiet waren schon dabei. All diese bislang untersuchten Proben enthalten Viren aus der sogenannten B.1 Lineage (Abstammungslinie) und deren Unterklasse B.1.1. Die Lineage B.1.1 ist bisher fast überall auf der Welt gefunden worden, hauptsächlich aber in Europa (siehe Karte). Weltweit sind knapp 100 SARS-CoV-2 Lineages beschrieben. »Die B.1.1 Lineage hat ihren Ursprung nach aktuellen Erkentnissen in der Lineage B.1 aus China, von der sie sich nur in wenigen Nukleotiden unterscheidet«, so Brandt. Die Zahlen hinter dem Buchstaben B geben die Mutationsvarianten an.
Doktorand Mike Marquet (v. r. n. l.), Dr. Christian Brandt und Dr. Oliwia Makarewicz vom Forschungslabor Klinische Infektiologie des Instituts für Infektionsmedizin und Krankenhaushygiene bei einer SARS-CoV-2 Sequenzierung mit dem MinION-System.
Foto: Michael Szabó/UKJZutritt für Viren verweigert
Bioinformatikerinnen und Bioinformatiker des Universitätsklinikums sind an einem Forschungsprojekt beteiligt, dass zum Ziel hat, Wirkstoffe zu entwickeln, die es dem Coronavirus unmöglich machen, sich in menschlichen Zellen zu vermehren. »Damit ließe sich eine Infektion effektiv stoppen«, sagt Prof. Dr. Rainer König von der Arbeitsgruppe Systembiologie der Sepsis, der das Teilprojekt am UKJ leitet. Denn: »Um sich zu vermehren, braucht das Virus die Reproduktionsmaschinerie von Wirtszellen.« Wie andere Viren auch, dringt SARS-CoV-2 in Zellen eines Wirtsorganismus ein, wird dort tausendfach kopiert, um anschließend weitere Zellen zu kapern und sich weiter zu vermehren. Die infizierten Zellen des Wirtes überleben das in der Regel nicht.
Prof. Rainer König
Foto: Anne Günther (Universität Jena)Anstatt nun Wirkstoffe gegen die sich schnell verändernden Virusproteine selbst zu entwickeln — was potenziell rasch zu Resistenzen führt —, verfolgt das Forschungsteam den Ansatz, die Proteine der Wirtszellen so mit Wirkstoffen zu blockieren, dass es den Viren unmöglich wird, sie zur Vermehrung zu nutzen. Dafür liegen bereits gut untersuchte Beispiele vor, wie etwa der unter dem Namen Maraviroc vertriebene Wirkstoff, der verhindert, das HI-Viren in Immunzellen eindringen können, indem der dafür notwendige Rezeptor blockiert wird.
In dem vom Bundesministerium für Bildung und Forschung mit rund 500.000 Euro finanzierten Projekt möchte das Konsortium um Prof. König, Prof. Dr. Sandra Ciesek (Universitätsklinikum Frankfurt/M.) und Dr. Holger Erfle (Universität Heidelberg) insgesamt etwa 10.000 Gene, für deren Proteine inhibierende Wirkstoffe entwickelt werden können oder schon entwickelt sind, einzeln herunter regulieren. In Hochdurchsatz-Screeningverfahren wollen die Forschenden messen, ob sich dadurch die Virusvermehrung verringern oder sogar stoppen lässt, sodass die Wirtszellen vor einer Infektion geschützt sind. Dafür werden Zellen einer menschlichen Modellzelllinie (CaCo-2) in Mikrotiterplatten angezüchtet und definierte Zellproteine einzeln ausgeschaltet. Anschließend wird versucht, die so behandelten Zellen mit Coronaviren zu infizieren. Anhand automatisiert aufgenommener mikroskopischer Aufnahmen lässt sich schließlich ablesen, bei welchen Zellen ein Schutzeffekt entstanden ist.
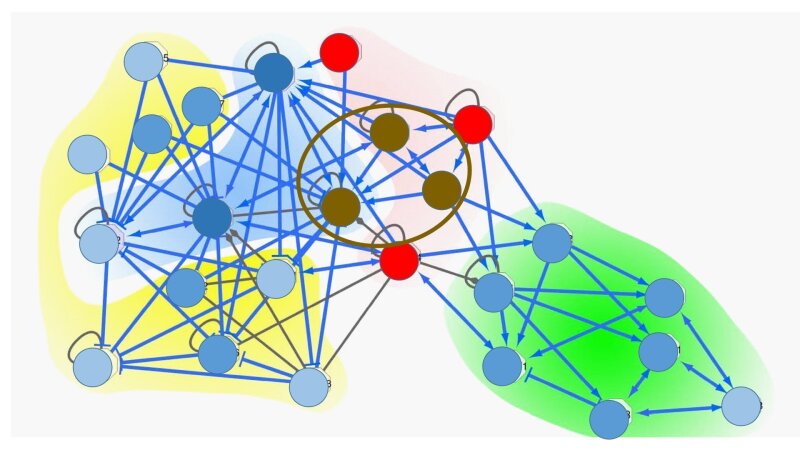
Die Bilder und Daten fließen dann in der Jenaer Arbeitsgruppe von Prof. König in eine zelluläre Netzwerkanalyse zusammen (siehe Abbildung). »Anhand dieser und weiterer molekularer Daten aus öffentlich zugänglichen Datenbanken wollen wir die zellbiologischen Prozesse identifizieren, die für den Lebenszyklus des Virus unverzichtbar sind.« Seien diese bekannt, lassen sich genau die passenden Wirkstoffe finden, die die Vermehrungsprozesse von SARS-CoV-2 am effektivsten stören.
Proteine, deren herunter geregelte Gene einen Schutzeffekt zeigen (hier schematisch in rot dargestellt), werden auf einem zellulären Netzwerk abgebildet, sogenannte Repressormodule definiert (rot unterlegt), und Wirkstoffe identifiziert, deren Targets mit diesen Modulen überlappen (braun).
Grafik: Rainer König»Wir können nicht ewig die Luft anhalten«
»Flatten the curve«, das war das Motto zu Beginn der Corona-Pandemie. Wie in anderen Ländern auch, haben Bund und Länder in Deutschland weitreichende Maßnahmen beschlossen, um die Dynamik des COVID-19-Ausbruchs zu verlangsamen. »Wichtig war es, Zeit zu gewinnen, um sich auf die Krankheit einzustellen«, sagt PD Dr. Dr. Petra Dickmann von der Klinik für Anästhesiologie und Intensivmedizin des Uniklinikums. Die zwischenzeitlich gesunkenen Fallzahlen in Deutschland zeigen, dass diese Strategie richtig war. »Doch Maßnahmen wie Schulschließung und Kontaktverbote sind natürlich nicht nachhaltig«, so Dickmann weiter. Schließlich könne man nicht ewig die »Luft anhalten« und müsse das öffentliche Leben wieder zulassen. Doch wie schon beim Herunterfahren des öffentlichen Lebens agieren die politisch Verantwortlichen derzeit auch bei der Lockerung der Maßnahmen weitgehend »auf Sicht«. »Es fehlen rationale Kriterien, wann und wie welche Maßnahmen gelockert werden können, ohne eine Überbeanspruchung des Gesundheitssystems zu erzeugen.«
Plakataktion in Jena während der Corona-Pandemie im März 2020.
Foto: Anne Günther (Universität Jena)Genau diese Kriterien wollen Petra Dickmann und ihr Team gemeinsam mit Kolleginnen und Kollegen im Rahmen der Forschungsgruppe »Pandemiemanagement« entwickeln und einen wissenschaftlich fundierten Background liefern, wie das Land geordnet aus der Pandemiesituation wieder herauskommt.
Das Projekt steht auf drei Säulen. Erstens zielt es auf ein besseres Verständnis der Krankheitslast. In diesem Zusammenhang hat die Forschungsgruppe schon ein erstes Konzept entwickelt: »Wir haben ein Frühwarnsystem vorgeschlagen, mit dem sich erfassen lässt, wie das Infektionsgeschehen aktuell aussieht«, sagt Petra Dickmann. Dafür sollen biostatistisch-sinnvoll regelmäßig Personen an sensiblen Orten wie etwa in Schulen, Alten- und Pflegeeinrichtungen sowie in Hotspots des Infektionsgeschehens auf das Coronavirus getestet werden. »Wir müssen herausfinden, wie viele Personen infiziert sind, ohne Symptome zu zeigen und so den Ausbruch vorantreiben könnten.« Die Forschenden verfolgen damit einen Testansatz, der die Infektionsrate in einer bestimmten Gruppe als Spiegelbild der Gesamtbevölkerung misst.
Zweitens geht es in dem Forschungsvorhaben darum, Kapazitäten und wechselnden Bedarf im Gesundheitssystem möglichst aktuell abzubilden. Um Corona-Patienten bestmöglich versorgen zu können, sind in den Kliniken Intensivbetten vorgehalten und zusätzliche Beatmungsgeräte gekauft worden. Doch was bedeutet diese Fokussierung auf COVID-19 für die Versorgungsmöglichkeiten aller anderen Patienten? Welche Untersuchungen und Behandlungen — vom Impftermin über Vorsorgeuntersuchungen bis zur Chemotherapie — finden aktuell statt und wie ist das Gesundheitssystem dafür gerüstet? Dazu hat die Forschungsgruppe an dem Intensivkonzept mitgearbeitet, das auf Landesebene die Rationale vorgibt.
Und drittens untersuchen Petra Dickmann und die beteiligten Arbeitsgruppen die Pandemiefolgen für die Bevölkerung. In Kooperation mit der COSMO Studie der Uni ErfurtExterner Link etwa werden Menschen danach befragt, wie sie die Pandemie und die Präventionsmaßnahmen in Thüringen wahrnehmen. In einem weiteren Strang wird die Risikokommunikation im Pandemiemanagement erforscht. Hier werden im Rahmen der »Neustadt-Studie« Erkenntnisse über die Akzeptanz von Quarantänemaßnahmen und die Informationsbedürfnisse der Menschen in Quarantänegebieten gewonnen. Diese Einsichten haben aktuellen Bezug auf die Forschungs- und politische Landschaft des Pandemiemanagements in Deutschland.
»Die Neustadt-Studie« — Keine Antikörper bei der Hälfte der Infizierten
Anfang März hatte die Corona-Pandemie auch Thüringen erreicht. Nach der Bestätigung der ersten Infektion mit dem neuartigen Virus am 2. März stieg die Zahl der nachgewiesenen Fälle kontinuierlich an. Hotspots des Krankheitsausbruches im Freistaat waren jedoch nicht große Städte wie Erfurt oder Jena — sondern unter anderem der knapp 900-Seelen-Ort Neustadt am Rennsteig. Hier infizierten sich in kurzer Zeit so viele Einwohner, dass der ganze Ort für zwei Wochen unter Quarantäne gestellt wurde. Am Ende der Quarantänezeit waren durch ein obligatorisches Screening der gesamten Bevölkerung 49 Corona-Infektionen bestätigt, zwei Patienten an COVID-19 verstorben. Hochgerechnet auf die Einwohnerzahl des Ortes lagen die Werte um ein Vielfaches höher als im landesweiten Durchschnitt.
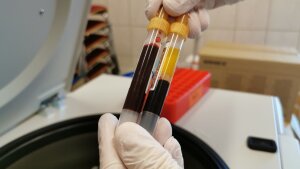
Blutproben von Einwohnern des Ortes Neustadt am Rennsteig in Thüringen, die im Rahmen der »Neustadt-Studie« am UKJ untersucht wurden. In dem Ort waren sehr viele COVID-19-Erkrankungen aufgetreten und alle Einwohner wurden unter Quarantäne gestellt.
Foto: UKJ/HoppertInzwischen hat sich die Situation in Neustadt, wie in den meisten Städten und Gemeinden in Deutschland, entspannt — Gelegenheit für Wissenschaftlerinnen und Wissenschaftler des Jenaer Universitätsklinikums den Ausbruch von Neustadt detailiert unter die Lupe zu nehmen. Ein zehnköpfiges Team unter Leitung von Prof. Dr. Mathias Pletz, dem Direktor des Instituts für Infektionsmedizin und Krankenhaushygiene, untersucht derzeit, inwiefern es zusätzlich zu den bereits bekannten 49 Fällen auch nicht bemerkte, asymptomatische Verläufe gab und, ob sich unter den Einwohnern von Neustadt eine Immunität ausgebildet hat. Dafür haben die Jenaer Forschenden von insgesamt 626 Bewohnern (das sind 71 Prozent aller Einwohner) aller Altersgruppen, darunter auch junge Kleinkinder, Blut- und Rachenspülwasserproben gesammelt, sie über Symptome während der Erkrankung und mögliche Kontakte mit dem Virus befragt.
Erste Ergebnisse der »Neustadt-Studie«pdf, 1 mb, die vom Freistaat Thüringen mit 500.000 Euro finanziert wird, liegen jetzt vor. Die Untersuchung ergab unter anderem, dass niemand im Ort aktuell positiv auf SARS-CoV-2 getestet wurde. »Im Rachenspülwasser konnten wir kein zirkulierendes Virus mehr nachweisen«, berichtet PD Dr. Sebastian Weis, der maßgeblich an der Planung und Durchführung der Studie beteiligt war. Bei etwa acht Prozent der untersuchten Personen wurden in mindestens zwei von insgesamt sechs verschiedenen COVID-19-Antikörpertests, sogenannte IgG-Antikörper gegen das Virus nachgewiesen. »Bei etwa der Hälfte der Studienteilnehmer, bei denen das Virus im März bestätigt worden war, fanden wir keine Antikörper – obwohl wir mehrere verschiedene Tests verwendet hatten«, so Prof. Mathias Pletz. Ob bei diesen Personen statt Antikörper spezielle Abwehrzellen gebildet wurden, wird gerade in weiteren Untersuchungen getestet. Ergebnisse dazu werden im Laufe des Sommers erwartet. Ein weiteres Ergebnis: Die Forschenden fanden auch Antikörper bei Personen, die nicht wissentlich mit SARS-CoV-2 infiziert waren. Ob diese Personen eine Immunität gegen das Virus aufgebaut haben, lasse sich aus den bisher vorliegenden Ergebnissen aber nicht sicher ableiten.
Neben dem Institut für Infektionsmedizin und Krankenhaushygiene sind vier weitere Insitute des UKJ an der Studie beteiligt.
COVID-19-Sprechstunde zu Hause
Vier von fünf mit dem neuartigen Coronavirus infizierten Patienten zeigen einen milden Krankheitsverlauf mit Symptomen, die denen einer schweren Erkältung ähneln: Fieber, Husten, Kopf- und Gliederschmerzen. »Diese Patienten können die Erkrankung in der Regel gut zu Hause auskurieren«, sagt PD Dr. Stefan Hagel vom Institut für Infektionsmedizin und Krankenhaushygiene des UKJ. Wichtig sei jedoch eine diagnostische Überwachung durch geschulte Mediziner, um eine mögliche Verschlechterung des Zustands festzustellen und gegebenenfalls eine stationäre Aufnahme einzuleiten. Denn: Wenn es kritisch wird, ist es für die Patienten überlebenswichtig, schnell zu handeln.
»Da sich die Patienten aber in häuslicher Quarantäne befinden, ist der Gang zum Hausarzt für sie keine Option«, so Dr. Hagel. Die meisten Ärzte vermeiden zudem Hausbesuche bei COVID-19-Patienten, um das Risiko einer weiteren Ausbreitung der Viren gering zu halten. Viele Betroffene fühlen sich mit der Krankheit allein gelassen. Eine Lösung für dieses Problem hat sich jetzt am Jenaer Uniklinikum etabliert. Hier kommt eine Handy-App zum Selbstmonitoring von Patienten zum Einsatz: Die »MedoPad-App« übermittelt mehrmals täglich diagnostische Parameter wie Sauerstoffsättigung im Blut, Herzfrequenz und Körpertemperatur des Patienten an die Klinik, wo Mediziner die Ergebnisse prüfen und so den Krankheitsverlauf der Betroffenen immer im Blick haben.
Sauerstoffmessung an der Fingerkuppe zur Überwachung von COVID-19-Patienten. Insgesamt 400 Sauerstoffmessgeräte stehen am Klinikum zur Verfügung und könnten an ambulante Patienten vergeben werden. Die Messwerte werden per App an das Klinikum gesendet.
Foto: Michael Szabó/UKJ»Wichtigstes Ziel ist es, schwere Verläufe zu vermeiden«, erläutert Mediziner Hagel. Wenn Patienten eine Lungenentzündung entwickeln, werde ihr Zustand häufig schnell kritisch und sie werden zu spät ins Krankenhaus eingewiesen. Und genau das lasse sich mit der Monitoring-App vermeiden.
In der aktuell laufenden Pilotphase, an der auf Initiative des Bundesgesundheitsministeriums neben dem Uniklinikum Jena auch die Uniklinik Heidelberg beteiligt ist, werden die Patienten mit einem Sauerstoffmessgerät ausgestattet, mit dem sie ihre Werte dreimal täglich messen. Die Daten werden von der App direkt an das Klinikum übermittelt. Sobald ein oder mehrere Werte eine kritische Grenze über- bzw. unterschreiten, nimmt der Arzt Kontakt zum Patienten auf und bespricht mit ihm die weitere Behandlung.
Das Feedback, so zieht Dr. Hagel nach zwölf Wochen mit der MedoPad-App eine erste Bilanz, ist sehr positiv. Allerdings ist die Zahl der Patienten, die die App bisher tatsächlich genutzt hat, noch überschaubar – glücklicherweise sind die Fallzahlen in Jena und Thüringen im Moment niedrig. Doch für eine mögliche zweite Infektionswelle sei man gut gerüstet. Insgesamt 400 Sauerstoffmessgeräte stehen zur Verfügung und könnten an ambulante Patienten vergeben werden.
PD Dr. Stefan Hagel und Dr. Aurelia Kimmig vom Institut für Infektionsmedizin und Krankenhaushygiene des UKJ werten Patientendaten aus, die über die MedoPad-App übermittelt wurden.
Foto: Michael Szabó/UKJ
Text: Ute Schönfelder
Mehr Informationen und weitere Forschungsprojekte zu COVID-19 der Jenaer Universitätsmedizin sind zu finden unter: https://www.uniklinikum-jena.de/Forschung/Covid_19_Forschung/Taskforce.htmlExterner Link.